
天然产物的结构具有高度多样性,根源在于其生物合成酶的功能可塑性。尽管已有研究揭示初级代谢酶可被招募至次级代谢途径,然而,多酶复合体如何实现功能转变,其具体的分子机制仍缺少直接的实验证据。
2026年1月31日,深圳湾实验室化学生物学研究所唐啸宇课题组与合作者在国际期刊Nature Communications发表题为“Evolutionary repurposing of a metabolic thiolase complex enables antibiotic biosynthesis”的研究论文。
该研究聚焦于一类由硫解酶家族成员——HMG-CoA synthase (HMGS)和acetyl-CoA acetyltransferase (ACAT),以及DUF35蛋白组成的多酶复合体。该复合体在古菌中参与甲羟戊酸(mevalonate)合成,是典型的初级代谢酶。然而,该研究发现,在链球菌中HMGS、ACAT和DUF35蛋白组成的硫解酶复体在保守结构框架上通过活性口袋重构,获得了催化非经典的傅-克酰基化反应的新功能,从而完成抗生素reutericyclin A生物合成的关键步骤。

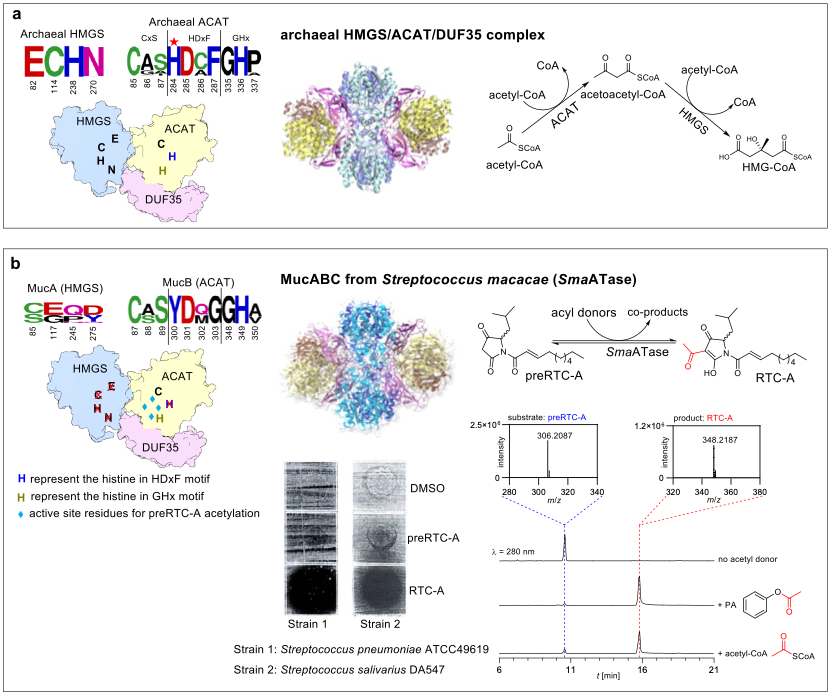
硫解酶复合体在保守结构框架内的功能创新;a古菌HMGS/ACAT/DUF35复合体催化连续两步Claisen缩合反应合成HMG-CoA;b链球菌中的MucABC多酶复合体(SmaATase)通过活性口袋重建,以preRTC-A为底物合成抗生素reutericyclin A
研究团队通过生化实验、冷冻电镜结构解析及系统的进化分析发现:
1. 该酶复合体在整体框架上仍高度保守,保持了典型的十二聚体装配方式;2. 其关键催化基序(如HMGS的E–C–H–N催化四联体、ACAT的HDxF基序)已发生退化或丢失;3. 活性位点经过精细重塑,使其不再催化Claisen缩合反应,而是专一识别小分子化合物preRTC-A并催化C–C键形成;4. 系统地生信分析结果表明,类似的HMGS/ACAT/DUF35基因组合在细菌中广泛存在,其中仅少数被真正招募进入次级代谢通路。
进一步活性研究表明,乙酰化修饰对reutericyclin A的抗菌活性至关重要:其未乙酰化前体分子的抑菌能力显著丧失。该结果提示该酶复合体的功能进化可能赋予了宿主菌在微生态竞争中的生存优势。
该研究不仅揭示了自然界中生物合成酶在保守结构框架内实现功能创新的进化策略,更为深入理解天然产物生物合成酶的演化规律,以及未来定向设计C–C键形成生物催化剂提供了重要理论依据。
本研究通讯作者为深圳湾实验室唐啸宇研究员、深圳医学科学院潘孝敬研究员、深圳湾实验室启航学者许建坤博士。深圳湾实验室廖格副研究员、西湖大学与医科院联培博士生孙若兰、深圳医学科学院博士后沈子琳为论文共同第一作者。香港大学李泳新教授也为本工作提供了重要支持。该研究得到了国家自然科学基金、广东省重点领域研发项目、深圳市重大科技专项、深圳湾实验室重点项目等多个项目的支持。
原文信息:
Evolutionary repurposing of a metabolic thiolase complex enables antibiotic biosynthesis
文章来源|唐啸宇课题组
编辑|鲍 啦
责编|远 山
欢迎投稿、建议|media@szbl.ac.cn

 粤公网安备44031102000926号
粤公网安备44031102000926号